Wasser ist aktives Bauelement in Proteinen. Wasser ist eine Protonendiode
Bochumer Biophysiker haben eine Diode für Protonen entdeckt: Genau wie das elektronische Bauteil die Flussrichtung des elektrischen Stroms vorgibt, sorgt die „Protonendiode“ dafür, dass Protonen nur in eine Richtung durch eine Zellmembran geschleust werden können. Wassermoleküle spielen dabei als aktive Bauteile der Diode eine wichtige Rolle. Die Forscher um Professor Klaus Gerwert (Lehrstuhl für Biophysik) konnten das durch eine Kombination aus Molekularbiologie, Röntgenstrukturanalyse, zeitaufgelöster FTIR-Spektroskopie und Biomolekularen Simulationen beobachten.
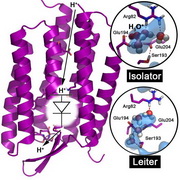
Die Protonendiode spielt eine wichtige Rolle bei der Energiegewinnung von Zellen. Lichtgetriebene Protonenpumpen – bestimmte Proteine, die die Zellmembran durchspannen – schleusen Protonen aus der Zelle heraus, sodass außen ein Überdruck entsteht, „ganz ähnlich wie der Wasserdruck an einer Staumauer“, verdeutlicht Gerwert. An anderer Stelle drängen die Protonen wieder in die Zellen hinein um das Konzentrationsgefälle auszugleichen, und treiben dabei die Turbinen der Zelle an, Proteine namens ATP-asen. Die dabei freiwerdende Energie wird umgewandelt in den universellen Kraftstoff der Zellen, ATP (Adenosintriphosphat). „Dieser Ablauf ist eine Art archaische Photosynthese“, erklärt Gerwert. „Die Lichtenergie wird letztlich in für den Organismus nutzbare Energie umgewandelt.“
Die Details dieser Vorgänge sind Gegenstand der Forschung. Besonders die Rolle der Wassermoleküle in Proteinen war lange unklar. „Früher glaubte man, dass die Wassermoleküle durch Zufall in Proteine hineingeraten würden und keine besondere Funktion erfüllten“, so Gerwert. Die aktuelle Arbeit zeigt, dass die Wassermoleküle im Protein sehr wohl eine aktive Funktion ausüben.
Die Bochumer Forscher konnten ihre Ergebnisse in einem interdisziplinären Ansatz durch eine Kombination aus Molekularbiologie, Röntgenstrukturanalyse, zeitaufgelöster FTIR-Spektroskopie und Biomolekularen Simulationen erzielen. Diese Kombination zeigt die dynamischen Vorgänge im Protein nach Lichtanregung mit atomarer Auflösung. „Man kann verfolgen, wie das Proton von der zentralen Protonenbindestelle im Innern des Proteins über eine Aminosäure und dann über einen protonierten Wassercluster an die Membranoberfläche transportiert wird“, beschreibt Gerwert. Der Ansatz erweitert jetzt die klassischen Methoden der Strukturbiologie, Röntgenstrukturanalyse und Kernspinresonanzspektroskopie (NMR), da er einen kompletten Film liefert und nicht nur Standbilder von Proteinen.
Quelle: Ruhr-Universität Bochum